4000. Proteinstruktur an BESSY II entschlüsselt
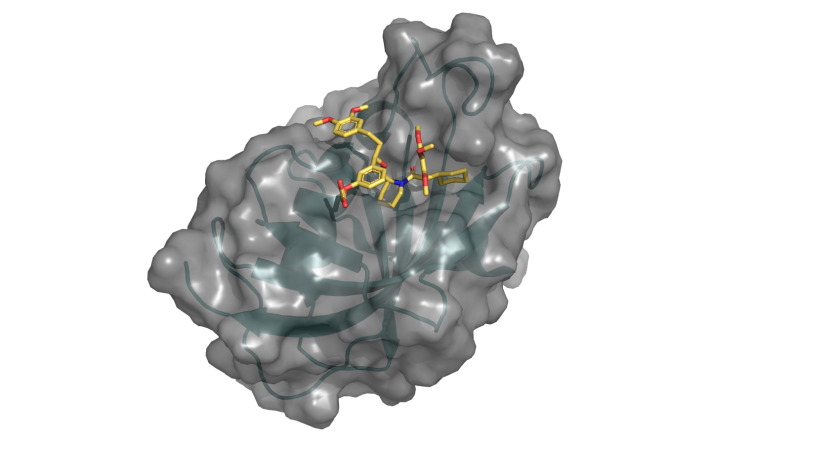
Die 4000. Proteinstruktur aus dem HZB BESSY, die in der PDB veröffentlicht wurde, zeigt die G64S-Variante von FKBP51 im Komplex mit dem hochselektiven Liganden SAFit (eingezeichnete Struktur). © C. Meyners/TU Darmstadt/HZB
Bei der 4000. Struktur handelt es sich um das Molekül FKBP51, das mit stressinduzierten Erkrankungen wie Depressionen, chronischen Schmerzen und Diabetes zusammenhängt. Das Team um Prof. Dr. Felix Hausch, TU Darmstadt, nutzt die Kenntnis der dreidimensionalen Struktur, um neue Strategien für das Design passender Medikamente zu entwickeln.
Viele Erkrankungen hängen mit Fehlfunktionen von Proteinen im Organismus zusammen. Die dreidimensionale Architektur dieser Moleküle ist oft äußerst komplex, liefert aber wertvolle Hinweise, wie sich die Fehlfunktion beheben ließe, beispielsweise durch Medikamente, die sich perfekt in eine „Tasche“ an das Zielmolekül binden und die Fehlfunktion blockieren. Die Struktur von Proteinen lässt sich mit Röntgenanalysen an den MX-Beamlines von BESSY II entschlüsseln.
In der Protein Data Bank (www.rcsb.org/pdb), die alle experimentell bestimmten Proteinstrukturen enthält, ist nun die 4000. Struktur aus BESSY II eingetragen worden. Das Team um Prof. Felix Hausch von der TU Darmstadt hatte Proteinkristalle aus dem Molekül FKBP51 hergestellt und an den MX-Beamlines untersucht.
Dabei handelt es sich um ein Protein, das eine besondere Rolle bei den großen Gesundheitsproblemen unserer Zeit spielt. Denn FKBP51 reguliert die Signaltransduktion von Steroidhormonrezeptoren, die durch Stress gestört sein kann. Dies kann Depressionen, chronische Schmerzen oder Krankheiten wie Diabetes und Übergewicht auslösen. Das Protein FKBP51 hat sich als vielversprechender Angriffspunkt für Medikamente gegen diese Krankheiten erwiesen. „Die Proteinstrukturanalyse zeigt uns, wo im Molekül interessante „Taschen“ sitzen, die als Zielpunkte für Medikamente in Frage kommen“, sagt Dr. Christian Meyners, TU Darmstadt.
arö
https://www.helmholtz-berlin.de/pubbin/news_seite?nid=24367;sprache=de
- Link kopieren
-
Zukunft der Korallen – Was Röntgenuntersuchungen zeigen können
In diesem Sommer war es in allen Medien. Angetrieben durch die Klimakrise haben nun auch die Ozeane einen kritischen Punkt überschritten, sie versauern immer mehr. Meeresschnecken zeigen bereits erste Schäden, aber die zunehmende Versauerung könnte auch die kalkhaltigen Skelettstrukturen von Korallen beeinträchtigen. Dabei leiden Korallen außerdem unter marinen Hitzewellen und Verschmutzung, die weltweit zur Korallenbleiche und zum Absterben ganzer Riffe führen. Wie genau wirkt sich die Versauerung auf die Skelettbildung aus?
Die Meeresbiologin Prof. Dr. Tali Mass von der Universität Haifa, Israel, ist Expertin für Steinkorallen. Zusammen mit Prof. Dr. Paul Zaslansky, Experte für Röntgenbildgebung an der Charité Berlin, untersuchte sie an BESSY II die Skelettbildung bei Babykorallen, die unter verschiedenen pH-Bedingungen aufgezogen wurden. Antonia Rötger befragte die beiden Experten online zu ihrer aktuellen Studie und der Zukunft der Korallenriffe.
-
Was vibrierende Moleküle über die Zellbiologie verraten
Mit Infrarot-Vibrationsspektroskopie an BESSY II lassen sich hochaufgelöste Karten von Molekülen in lebenden Zellen und Zellorganellen in ihrer natürlichen wässrigen Umgebung erstellen, zeigt eine neue Studie von einem Team aus HZB und Humboldt-Universität zu Berlin. Die Nano-IR-Spektroskopie mit SNOM an der IRIS-Beamline eignet sich, um winzige biologische Proben zu untersuchen und Infrarotbilder der Molekülschwingungen mit Nanometer-Auflösung zu erzeugen. Es ist sogar möglich, 3D-Informationen, also Infrarot-Tomogramme, aufzuzeichnen. Um das Verfahren zu testen, hat das Team Fibroblasten auf einer hochtransparenten SiC-Membran gezüchtet und in vivo untersucht. Die Methode ermöglicht neue Einblicke in die Zellbiologie.
-
Iridiumfreie Katalysatoren für die saure Wasserelektrolyse untersucht
Wasserstoff wird künftig eine wichtige Rolle spielen, als Brennstoff und als Rohstoff für die Industrie. Um jedoch relevante Mengen an Wasserstoff zu produzieren, muss Wasserelektrolyse im Multi-Gigawatt-Maßstab machbar werden. Ein Engpass sind die benötigten Katalysatoren, insbesondere Iridium ist ein extrem seltenes Element. Eine internationale Kooperation hat daher Iridiumfreie Katalysatoren für die saure Wasserelektrolyse untersucht, die auf dem Element Kobalt basieren. Durch Untersuchungen, unter anderem am LiXEdrom an der Berliner Röntgenquelle BESSY II, konnten sie Prozesse bei der Wasserelektrolyse in einem Kobalt-Eisen-Blei-Oxid-Material als Anode aufklären. Die Studie ist in Nature Energy publiziert.
